Nature: 基因研究揭示东南亚大陆复杂的人群遗传结构和演化历史
期刊:Nature
英文题目:Genome diversity and signatures of naturalselection in mainland Southeast Asia
中文题目:东南亚大陆人群基因组多样性和自然选择特征
作者(前三):Yaoxi He, Xiaoming Zhang, Min-Sheng Peng.
发表日期:2025年5月14日
摘要
东南亚大陆(Mainland Southeast Asia,MSEA)拥有近3亿人口,具有丰富的民族和文化多样性,然而在当前人类基因组数据库中,东南亚大陆的人群却鲜有代表性。为此,研究团队发布了包含3023名来自东南亚大陆30个不同族群的个体的短读全基因组测序数据,以及37名代表性个体的长读全基因组测序数据的SEA3K基因组数据集(第一阶段)。本研究从中识别出7959万个小型变异位点和96384个结构变异位点,其中有2283万个小型变异位点和24622个结构变异位点是该数据集中独有的。本研究观察到东南亚各人群之间存在高度的遗传异质性,这反映在遗传成分组合的多样性上。同时,识别出44个具有强烈达尔文正选择信号的基因组区域,涵盖89个基因,这些基因涉及多种生理系统,如身体特征和免疫反应等。此外,研究发现东南亚人群中存在不同的古老丹尼索瓦人基因流入,支持了丹尼索瓦人基因至少两次独立进入亚洲现代人类的假说。本研究还检测到东南亚人群中可能存在适应性古老基因流入的基因组区域。东南亚人群中大量新的基因组变异凸显了对区域人群开展研究的必要性,这有助于回答与史前历史、遗传适应和复杂疾病相关的关键问题。
研究背景
东南亚是全球最重要的人类演化区域之一,该地区人群拥有极高的遗传多样性,然而该地区的基因组学研究长期缺失,严重制约了人类环境适应性进化与疾病遗传机制的深度解析,因而也被称为全球人类基因组研究“最后一块拼图”。
由中国科学院昆明动物研究所牵头、泰国、柬埔寨等东南亚六国34个科研团队。深入东南亚大陆雨林腹地,采集了涵盖五大语系、6个国家、30多个地方的东南亚人群样本,完成了3023例深度全基因组测序。
本研究构建了迄今为止最完整的东南亚人群高质量全基因组变异数据库——SEA3K。研究发现大多数东南亚人群的遗传结构主要与他们的地理分布格局一致,而非按语系聚类,表明人群间的分化主要通过地理隔离形成,印证了该地区复杂的遗传交融与语言更替历史;系统解析东南亚人群的基因组多样性,发现东南亚人群适应热带雨林环境的关键基因,这些基因涉及体质、免疫、代谢等适应性特征,揭示了东南亚人群应对热带环境的独特进化策略;研究团队利用SEA3K高精度人群变异位点数据,结合全球人群基因组数据,对古人类基因渗入进行了系统分析,证明东南亚人群中确实存在多种丹尼索瓦人渗入模式;同时鉴定出多个具有正向选择信号的古人类基因片段,涉及代谢调节、免疫增强及肌肉发育等关键功能,提示来自古人类的基因序列很可能为东南亚人群适应热带雨林环境产生了重要贡献。研究团队还通过对SEA3K数据进行系统注释,鉴定出10个东南亚特异高频的致病变异,为探索罕见病与遗传变异的关联提供了天然研究模型,对未来东南亚地区人群的疾病风险评估与遗传咨询具有重要意义。
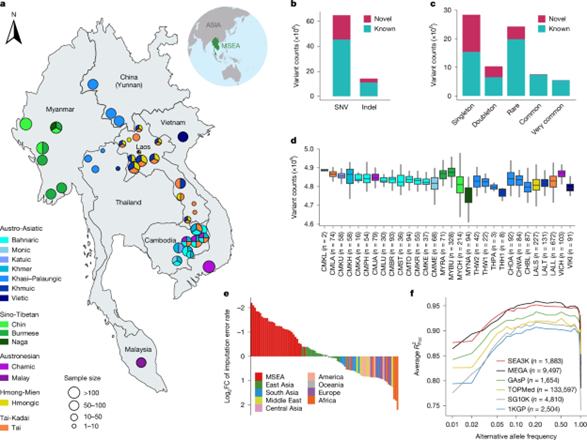
图1. SEA 3 K基因组变异的统计。a,选择的40个MSEA个体的地理位置用于长读序测序。饼图指示每个群体中所讲的语言的组成(参见图1a)。b,显示74个部分定相和组装的单倍型的组装邻接的虚线图。包括两个参考基因组(T2 T-CHM 13和GRCh 38(黑线))的重叠群用于比较。 c,SV发现示意图。使用Sniffles 2、PAV和SVision-pro进行基于读取映射(顶部)和基于汇编(底部)的调用,以调用简单SV和复杂SV。d,MSEA群体中每种SV类别的计数,包括缺失(DEL)、插入(INS)、重复(DUP)、倒位(INV)和复合SV(CSV)。e. 使用SEA3K参考面板对基因型推算性能进行评估。比较使用SEA3K面板和1KGP面板对全球各人群进行基因型推算时,推算错误率的倍数变化(FC)。补充图2展示了与其他面板的额外比较结果。f. 使用平方皮尔逊相关系数评估基因型推算的准确性,表明SEA3K面板相较于其他面板具有更优的性能。图中还标注了每个面板的样本量。MEGA面板整合了SEA3K、1KGP和SG10K数据集。目标人群是通过对2183名无亲缘关系的SEA3K个体随机抽取300名个体生成的,剩余的1883个样本用于进行单倍型定相(方法详见文中)。
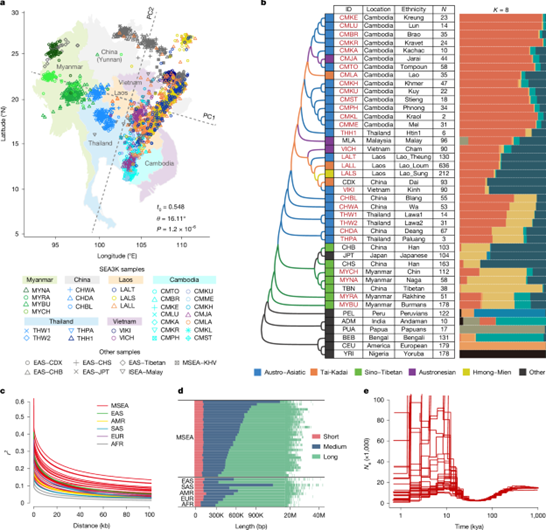
图2. 东南亚大陆(MSEA)人群的遗传结构和种群历史。a. 经过Procrustes变换的主成分分析(PCA)图,显示了2183名无关的MSEA个体的遗传差异与地理距离之间的高度相似性。Procrustes相似性统计量t0 = 0.548,基于10万次排列的P值为1.2×10⁻⁶(单侧排列检验),PCA图的旋转角度θ = 16.11°。虚线分别指示主成分1(PC1)和主成分2(PC2),它们分别解释了总变异的1.02%和0.53%。颜色表示本研究中新测序的人群。人群缩写详见补充表2。阴影背景指示样本的国家来源。ISEA表示岛屿东南亚;KHV表示越南胡志明市的京族人群。b. 最大似然树显示了MSEA(红色突出显示)与其他代表性全球人群之间的遗传关系,包括来自1KGP、HGDP、SGDP、马来人和藏族人群的代表性亚洲样本。混血图显示了SEA3K和参考人群(K=8)的遗传成分。c. 不同人群间的连锁不平衡(LD)衰减比较。参考了来自1KGP的五个超人群(每个超人群包含两个代表性人群)。AFR表示非洲人群;AMR表示美洲人群;EAS表示东亚人群;EUR表示欧洲人群;SAS表示南亚人群。d. 按大小类别(短、中、长)排序并着色的ROH(纯合子连续区段)分布,表明与1KGP人群相比,SEA3K人群的中等和长ROH类别边界向更长的ROH方向移动(选择了五个超人群的每组两个代表性人群进行比较,详情见补充表9)。K表示千;M表示百万。e. 推测的有效种群大小(Ne)随时间的变化,表明MSEA各人群的种群动态存在差异。使用成对连续MSMC2方法估计有效种群大小。分层种群版本和SMC++的结果展示在扩展数据图6中。
研究意义
这是一项由中国科学院昆明动物研究所牵头、泰国、柬埔寨等东南亚六国34个科研团队参与的国际合作研究在《自然》发表。该研究不仅极大地丰富了东南亚人群的基因组数据资源,还为深入探究该地区人群的史前迁徙、遗传适应以及复杂疾病提供了宝贵的基因组学依据。研究中发现的大量新型基因组变异强调了对区域人群开展研究的必要性,这些研究成果将有助于解答与东南亚人群相关的诸多关键科学问题,并为该地区精准医学的发展奠定基础。此外,研究团队构建的 SEA3K 基因组数据集及其相应的基因型推算参考面板,将为东南亚人群的遗传学研究提供重要的工具,有望提升对复杂性状和疾病的关联研究能力,进一步推动东南亚地区基因组学研究的进步。
创新之处
研究团队通过开展系统的基因组学、群体遗传学以及分子进化研究,发布了首个大规模的东南亚基因组数据集 SEA3K(第一阶段),这个数据集显著增加了东南亚人群基因组变异的代表性,为研究该地区人群的遗传多样性和适应性提供了宝贵的资源。研究揭示了东南亚人群中存在至少两次不同的 Denisovan 基因与现代人类的混合事件,并且发现了一些人群可能有更多次的混合事件。这种复杂的古人类基因流入模式为研究人类与其他古人类之间的基因交流提供了新的视角。同时,在东南亚人群中识别出44个具有强烈达尔文正选择信号的基因组区域,涵盖89个基因,这些基因涉及多种生理系统,如身体特征和免疫反应等。这些发现增进了对东南亚人群适应环境(如热带气候和病原体)的遗传机制的理解。该研究还通过对SEA3K数据集的分析,评估了东南亚人群中已知疾病风险变异的流行情况,并发现了一些在东南亚人群中常见的变异在其他地区是罕见的。这为未来在该地区开展精准医学研究和应用提供了重要信息。
对我们开展工作的启示
这篇发表在《自然》杂志上的文章通过对东南亚大陆(MSEA)3023名个体的深度基因组测序,构建了SEA3K基因组数据集,揭示了该地区人群的遗传多样性和适应性进化特征。科研工作者在研究中应重视开展广泛的跨区域、跨学科的交流与合作,重视科研数据的全面性和多样性,积极开发和利用大型数据集来推动科学发现。科研工作者应积极响应开放科学的理念,分享研究数据和资源,共同推动科学研究的发展。
文献来源:https://doi.org/10.1038/s41586-025-08998-w
声明:以上中文翻译为译者个人对于文章的概略理解,论文传递的准确信息请参照英文原文。
撰稿:邓艳青
初审:任杰
复审:杜军
终审:鲁鹏
